La frecuencia con la que normalmente se cometen errores durante la replicación del ADN es mínima. Sin embargo, hay situaciones en las que ocurren cambios en el genoma a una frecuencia mucho más alta de lo previsto, dando lugar a una patología celular cuyo resultado es la inestabilidad genética. Los estudios sobre Inestabilidad Genética han disfrutado en los últimos años de un gran interés en Biología Molecular y Biomedicina al ser una patología comúnmente asociada a las células cancerígenas y responsable de un número de enfermedades genéticas raras como la Xeroderma pigmentosum, los síndromes de Cockayne, de Werner, de Bloom, de Nijmegen, etc., todas ellas resultado del mal funcionamiento de los mecanismos de reparación del DNA y de respuesta al daño genotóxico y que presentan una fuerte incidencia de cáncer y/o envejecimiento prematuro. Una de las formas de inestabilidad con más impacto celular es la que se produce por recombinación del DNA, ya que puede dar lugar a reorganizaciones cromosómicas que conllevan la pérdida de heterocigosidad o la formación de genes quimeras que alteren el ciclo y la proliferación celular. Estos fenómenos de recombinación se suelen producir por la sobreacumulación de roturas cromosómicas de forma espontánea o por fallos en los mecanismos de reparación y señalización celular del daño (Aguilera y Gómez-González 2008).
Desde la década de los 80 se sabe que en células somáticas o en crecimiento vegetativo el DNA que se transcribe recombina más que el que no se transcribe. La interpretación actual de este hecho es que el DNA de los genes activos es más proclive a romperse que el DNA intergénico o de genes silenciados. Esto se ha visto desde bacterias a células humanas (Aguilera 2002, Kim and Jinks-Robertson 2012). ¿Por qué ocurre esto? La recombinación en células vegetativas resulta mayoritariamente de la reparación de roturas íntimamente ligadas al proceso de replicación. La replicación del DNA es un proceso en el que la célula corre altos riesgos, como los de incorporar nucleótidos erróneos o que se atasquen las horquillas de replicación y se rompan. La transcripción puede contribuir a este hecho ya que supone un obstáculo para la progresión de la horquilla. El problema es mayor entre polimerasas convergentes. El aumento de la inestabilidad de un gen al transcribirse solo se detecta si se transcribe durante la fase S del ciclo celular de forma simultánea con la replicación.
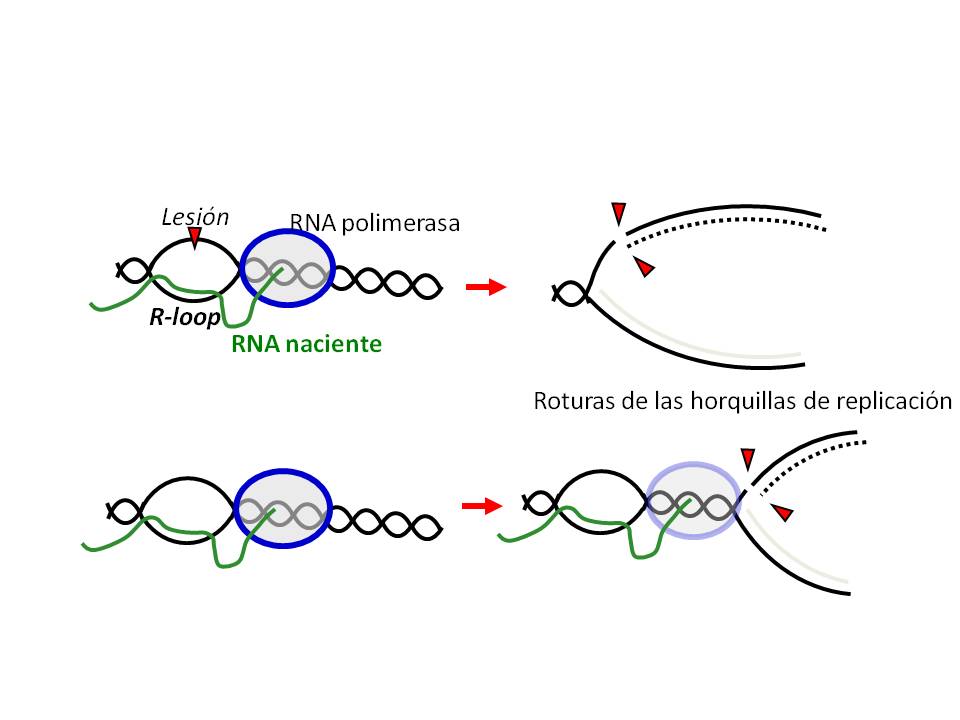
En este fenómeno, conocido como recombinación asociada a transcripción (TAR en inglés) un intermediario que puede jugar un papel relevante es el híbrido de DNA:RNA o R-loop (Huertas y Aguilera 2003). Este se forma como un producto colateral de la transcripción y las células disponen de una maquinaria específica, las ribonucleasas H, para eliminar el RNA de dichas estructuras. Durante la transcripción, el RNA naciente puede re-hibridar con su DNA molde aprovechando el superenrollamiento negativo que éste acumula transitoriamente por detrás de la RNA polimerasa elongante. Los R-loops juegan un papel esencial en la recombinación responsable del cambio de isotipo de las Inmunoglobulinas (Yu et al. 2003). No obstante, el propio empaquetamiento proteínico del RNA naciente para formar la ribonucleopartícula así como las maquinarias de procesamiento del RNA y transporte al citoplasma impiden que se formen esos híbridos. Entre estas proteínas se encuentran THO, Sub2/UAP56, THSC/TREX-2 o Sen1/SETX, conservadas desde levaduras a humanos (Luna et al. 2009). Cuando algunas de estas proteínas no funcionan correctamente se produce un fuerte fenómeno de inestabilidad genética debido a la acumulación de R-loops (Figura).
Recientemente, la recombinación asociada a transcripción y los híbridos de DNA:RNA han adquirido una relevancia insospechada. Numerosos trabajos han permitido demostrar que la conocida inestabilidad de las secuencias repetidas de tripletes y, muy especialmente los sitios frágiles, regiones cromosómicas que tienden a romperse con frecuencia, están asociados a regiones de ADN que se transcriben y a la formación de R-loops (Aguilera and García-Muse 2012). Es decir, existen secuencias de ADN proclives a romperse como consecuencia de su transcripción por una vía mediada por R-loops.
No sabemos aún por qué un R-loop tiene tendencia a formarse en unas regiones frente a otras, cómo genera inestabilidad, qué factores contribuyen a su formación, cómo afectan a la progresión de las horquillas de replicación o a la estabilidad de la RNA polimerasa, en qué se diferencia una recombinación asociada a R-loop o no asociada. Responder a estas cuestiones es clave para entender la dinámica de los genomas y muy especialmente cómo la biogénesis y procesamiento del RNA desde la transcripción al transporte núcleo-citoplasma pueden modular la integridad de los genomas.
Referencias:
- Aguilera A, Gómez-González B. 2008. Genome instability: a mechanistic view of its causes and consequences. Nat Rev Genet. 9:204-217.
- Kim N, Jinks-Robertson S. 2012. Transcription as a source of genome instability. Nat Rev Genet. 13:204-214.
- Huertas P, Aguilera A. 2003. Cotranscriptionally formed DNA:RNA hybrids mediate transcription elongation impairment and transcription-associated recombination. Mol. Cell 12:711-721.
- Yu K, Chedin F, Hsieh CL, Wilson TE, Lieber MR. 2003. R-loops at immunoglobulin class switch regions in the chromosomes of stimulated B cells. Nat Immunol. 4:442-451.
- Luna R, Gaillard H, González-Aguilera C, Aguilera A. 2008. Biogénesis of mRNPs: integrating different processes in the eukaryotic nucleus. Chromosoma 117:319-331.
- Aguilera A, García Muse T. 2012. R loops: from transcription byproducts to threats to genome stability. Mol. Cell 46:115-124.
